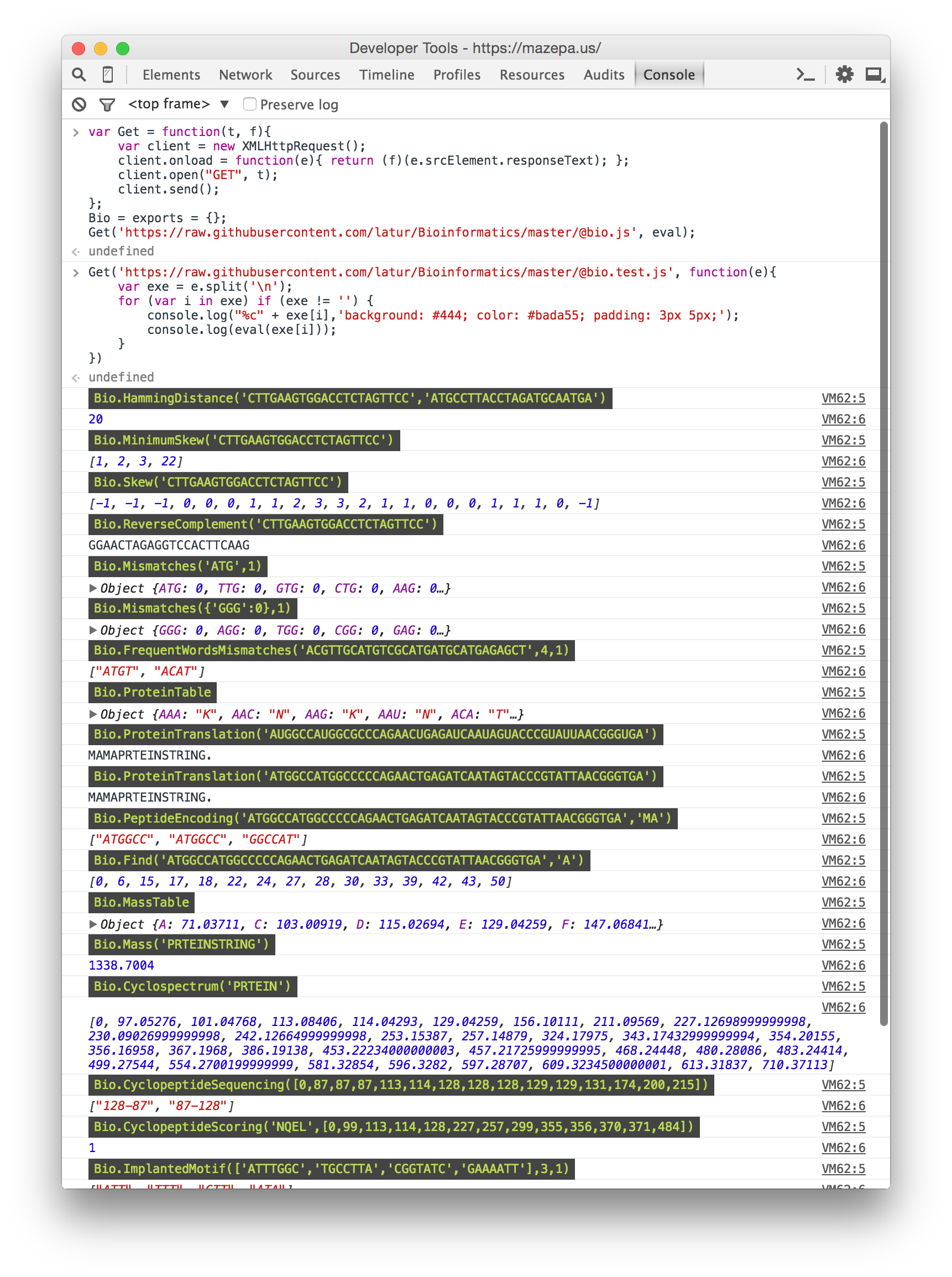
Быстрое решение биоинформатических задач в консоли браузера.
var Get = function(t, f){
var client = new XMLHttpRequest();
client.onload = function(e){ return (f)(e.srcElement.responseText); };
client.open("GET", t);
client.send();
};
Bio = exports = {};
Get('https://raw.githubusercontent.com/latur/Bioinformatics/master/@bio.js', eval)
Get('https://raw.githubusercontent.com/latur/Bioinformatics/master/@bio.test.js', function(e){
var exe = e.split('\n');
for (var i in exe) if (exe != '') {
console.log("%c" + exe[i],'background: #444; color: #bada55; padding: 3px 5px;');
console.log(eval(exe[i]));
}
})
Bio = require('./@bio.js');
Расстояние Хэмминга. Количество отличий в соответствующих последовательностей
Bio.HammingDistance('ATGC', 'ATGC');
Минимум перевеса нуклеотида G. Возращает все точки минимума
Bio.MinimumSkew('ATGC');
Нахождение последовательности, обратно-комплементарной к данной
Bio.ReverseComplement('ATGC')
Нахождение всех последовательностей, которые возможно получить из данной за d замен
Bio.Mismatches('ATGC', 2)
Нахождение встречающегося чаще других k-мера с точностью до d мутаций
Bio.FrequentWordsMismatches('ATGCATGC', 4, 1)
Таблица замен триплета ДНК на нуклеотид
Bio.ProteinTable
Получение из последовательности РНК пептидной
Bio.ProteinTranslation('AUGGCCAUGG');
Bio.ProteinTranslation('ATGGCCATGG');
Нахождение подпоследовательности, которая может кодировать данный пептид
Bio.PeptideEncoding('ATGGCCATG', 'MA')
Поиск подстроки в строке. Результат — массив точек вхождения
Bio.Find('ATGGATCCAGAACTG', 'A')
Таблица масс аминокислоты в дальтонах (округление до целых чисел)
Bio.MassTable
Масса пептида в дальтонах
Bio.Mass('PRTEINSTRING')
Теоретический спектр циклопептида
Bio.Cyclospectrum('PRTEIN')
Получение всех циклопептидов с данным спектром
Bio.CyclopeptideSequencing([0,87,87,87,113,114,128,128,128,129,129]);
Рейтинг спектра — число совпадений теоретического с экспериментальным
CyclopeptideScoring('NQEL', [0,99,113,114,128,227,257,299,355,356,370,371,484]);